天然無序蛋白(intrinsically disorder protein, IDP)廣泛存在于真核生物的蛋白質組中,并在遺傳信息轉錄、信號轉導等生命過程中發揮關鍵作用。如何對天然無序蛋白高度柔性和異質的構象進行高分辨刻畫一直是該領域方法學發展中的重點問題。超過15%的IDP分子在細胞內處于膜結合狀態,它們在磷脂雙分子層環境下的內部及全局運動(internal and global motions)與其理化性質和生物功能息息相關,但這些動態過程難以通過常規結構分析方法進行捕獲和定量表征。
近期,中國科學技術大學龍冬教授課題組發展了基于膜順磁弛豫增強技術(mPRE)的IDP波譜分析方法,成功實現對膜結合IDP分子內部構象動態、膜內旋轉及浸沒深度的高精度建模。相關成果以“Quantitative Ensemble Interpretation of Membrane Paramagnetic Relaxation Enhancement (mPRE) for Studying Membrane-Associated Intrinsically Disordered Proteins”為題發表于著名學術期刊《美國化學會志》(Journal of the American Chemical Society)。
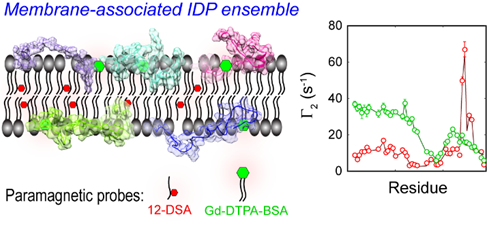
圖1. 基于mPRE順磁弛豫速率(Γ2)的膜結合態IDP系綜建模。
在該工作中,研究人員為準確解釋mPRE波譜數據,詳細探究了自旋探針分子在膜內的柔性和流動性,并提出加權三維點陣模型用于定量刻畫自旋探針動態性對膜順磁弛豫增強速率(Γ2)的影響。利用該模型的計算高效性,研究人員進一步發展了mPRE實驗數據引導的迭代更新算法,對膜結合天然無序蛋白的全局和內部運動自由度進行系統優化,實現了隱式膜環境下IDP全原子系綜模型的構建。作為原理驗證和應用示范,該方法被用于解析CD3ε胞質結構域(CD3εCD)在脂質雙膠束(lipid bicelle)中的動力學系綜。CD3εCD包含有重要的酪氨酸信號轉導基序(ITAM),以固有無序狀態與脂質雙分子層形成模糊復合物(fuzzy complex),并利用膜對關鍵酪氨酸位點的動態屏蔽來調節其信號轉導活性。基于mPRE實驗參數生成的動力學系綜在原子水平上詳細繪制了CD3εCD在膜內不同區域的動態分布,并揭示ITAM上不同酪氨酸位點與膜作用的關鍵差異,為T細胞中起抑制反饋作用的ITAM單磷酸化(monophosphorylation)狀態提供了新的機制性解釋。該工作所確立的mPRE波譜分析方法預期可廣泛推動各種功能性膜天然無序蛋白的原子分辨率研究。
該課題組博士研究生金紅、博士后柳丹為論文共同第一作者。該研究工作得到國家重點研發計劃、國家自然科學基金和中央高校基本科研業務費的資助。
論文鏈接:https://pubs.acs.org/doi/10.1021/jacs.3c10847
(生命科學與醫學部、科研部)

